胡红丽,博士,校长青年学者,胡红丽| 香港中文大学(深圳)医学院助理教授。
胡红丽博士主要从事 G 蛋白偶联受体(简称 GPCR)的复合物研究,运用冷冻电镜技术得到 GPCR 的高分辨结构,并研究膜蛋白与其调控蛋白之间相互作用,为设计以膜蛋白为靶标的药物提供重要的理论依据。自2017年起在 Nature、Cell 等顶级杂志上发表文章五篇,其中以共同第一作者身份在 Nature杂志发表文章两篇。主要成果包括:阿片受体与抑制型G蛋白Gi 的3.5埃高分辨电镜结构,以及首个C类GPCR代谢型谷氨酸受体(mGlu5)全长分子不同状态的电镜结构。两个成果均发表在Nature杂志上,胡红丽博士均为共同第一作者,两篇文章均被Nature News & Review着重报道。
教育及工作经历:
2013年获得北京大学医学部生物物理学专业博士学位。
2008年获得南开大学应用物理学专业学士学位。
分别在美国纽约州卫生部Wadsworth Center、密歇根大学和斯坦福大学从事基于冷冻电子显微镜的结构生物学的研究,其中在密歇根大学和斯坦福大学期间与2012 年诺贝尔化学奖得主 Brian K. Kobilka 教授和Robert J. Lefkowitz教授展开广泛的合作研究,参与了许多重要项目,在解析G 蛋白偶联受体高分辨的复合物结构方面做出了突出贡献。
2019年6月起入职香港中文大学(深圳),目前在医学院及科比尔卡创新药物研究院担任助理教授/独立PI、博士生导师
学术兼职:
资料更新中……
研究领域:
基于冷冻电子显微镜的结构生物学、膜蛋白结构与功能、G蛋白偶联受体结构及功能、药物筛选。
承担科研项目:
资料更新中……
科研成果:
资料更新中……
发明专利:
资料更新中……
发表学术论文:
1.Guo, Qiong; He, Binbin; Zhong, Yixuan; Jiao, Haizhan; Ren, Yinhang; Wang, Qinggong; Ge, Qiangqiang; Gao, Yongxiang; Liu, Xiangyu; Du, Yang; Hu, Hongli*; Tao, Yuyong*.A method for structure determination of GPCRs in various states.Nature Chemical Biology, 2024, 20(1): 74-+.
2.Jiao, Haizhan; Pang, Bin; Chiang, Ying-Chih; Chen, Qiang; Pan, Qi; Ren, Ruobing*; Hu, Hongli*.Structure basis for the modulation of CXC chemokine receptor 3 by antagonist AMG487.Cell Discovery, 2023, 9(1): 119.
3.Bing Gan#; Leiye Yu#; Haifeng Yang#; Haizhan Jiao; Bin Pang; Yian Chen; Chen Wang; Rui Lv; HU HONGLI; Zhijian Cao*; Ruobing Ren*.Mechanism of agonist-induced activation of the human itch receptor MRGPRX1.PLOS BIOLOGY, 2023, 21(6): e3001975.
4.Geng Chen#; Xiankun Wang#; Qiwen Liao#; Yunjun Ge#; Haizhan Jiao; Qiang Chen; Yezhou Liu; Wenping Lyu; Lizhe Zhu; Gydo C.P.van Zundert; Michael J.Robertson; Georgios Skiniotis; Yang Du*; HU HONGLI*; Richard D.Ye*.Structural basis for recognition of N-formyl peptides as pathogen-associated molecular patterns.Nature Communications, 2022, 13(1): 5232.
5.Yu Leiye#; He Licong#; Gan Bing#; Ti Rujuan#; Xiao Qingjie; Xin Yang*; Hu Hongli; Zhu Lizhe*; Wang Sheng*; Ren Ruobing*.Structural insights into sphingosine-1-phosphate receptor activation..Proceedings of the National Academy of Sciences of the United States of America, 2022, 119(16).
6.Liu, Dongmei; Chen, Qiang; Zhang, Lei; Hu, Hongli; Yin, Changcheng*.AgsA oligomer acts as a functional unit.Biochemical and Biophysical Research Communications, 2020, 530(1): 22-28.
7.Hilger, Daniel#; Kumar, Kaavya Krishna#; Hu, Hongli#; Pedersen, Mie Fabricius; O'Brien, Evan S.; Giehm, Lise; Jennings, Christine; Eskici, Gozde; Inoue, Asuka; Lerch, Michael; Mathiesen, Jesper Mosoliff*; Skiniotis, Georgios*; Kobilka, Brian K.*.Structural insights into differences inGprotein activation by family Aand family BGPCRs Science, 2020, 369(6503): 523-+..
8.Staus, Dean P.#; Hu, Hongli#; Robertson, Michael J.#; Kleinhenz, Alissa L. W.; Wingler, Laura M.; Capel, William D.; Latorraca, Naomi R.; Lefkowitz, Robert J.*; Skiniotis, Georgios*.Structure of the M2 muscarinic receptor-beta-arrestin complex in a lipid nanodisc.Nature, 2020, 579.
9.Yang Gao#; HU HONGLI#; Sekar Ramachandran#; Jon W. Erickson; Richard A. Cerione*; Georgios Skiniotis*.Structures of the Rhodopsin-Transducin Complex: Insights into G-Protein Activation.Molecular Cell, 2019, 75(1): 781-790.
10.Kato, Hideaki E; Zhang, Yan; Hu, Hongli; Suomivuori, Carl Mikael; Kadji, Francois Marie Ngako; Aoki, Junken; Kumar, Kaavya Krishna; Fonseca, Rasmus; Hilger, Daniel; Huang, Weijiao; Latorraca, Naomi R; Inoue, Asuka; Dror, Ron O; Kobilka, Brian K*; Skiniotis, Georgios*.Conformational transitions of a neurotensin receptor 1-G(i1) complex.Nature, 2019, 572(7767): 80-+.
11.Koehl, Antoine#; Hu, Hongli#; Feng, Dan#; Sun, Bingfa; Zhang, Yan; Robertson, Michael J.; Chu, Matthew; Kobilka, Tong Sun; Laermans, Toon; Steyaert, Jan; Tarrasch, Jeffrey; Dutta, Somnath; Fonseca, Rasmus; Weis, William I.; Mathiesen, Jesper M.*; Skiniotis, Georgios*; Kobilka, Brian K.*.Structural insights into the activation of metabotropic glutamate receptors.Nature, 2019, 566(7742): 79-+.
12. Antoine Koehl*, Hongli Hu*, Dan Feng*, Bingfa Sun, Yan Zhang, Matthew Chu, Tong Sun Kobilka, Els Pardon, Jan Staeyart, Jeffrey Tarrasch, Somnath Dutta, Rasmus Fonseca , William I Weis, Jesper Matthiesen, Georgios Skiniotis, Brian K Kobilka. "Structural Insights into Metabotropic Glutamate Receptor Activation". Nature, 566(2019):79–84
13. Kaavya Krishna Kumar*, Moran Shalev-Benami*, Michael J. Robertson, Hongli Hu, Samuel D. Banister, Scott A. Hollingsworth, Naomi R. Latorraca, Hideaki E. Kato, Daniel Hilger, Shoji Maeda, William I. Weis, David L. Farrens, Ron O. Dror, Sanjay V. Malhotra, Brian K. Kobilka, Georgios Skiniotis. “Structure of a Signaling Cannabinoid Receptor 1-G Protein Complex”, Cell (2019), 176 (3), 448-458. e12
14.Shoji Maeda; Antoine Koehl; Hugues Matile; Hongli Hu; Daniel Hilger; Gebhard F X Schertler; Aashish Manglik; Georgios Skiniotis; Roger J P Dawson; Brian K Kobilka.Development of an antibody fragment that stabilizes GPCR/G-protein complexes.Nature Communications, 2018, 9(1): 3712.
15.Qu Qianhui; Takahashi Yoh hei; Yang Yidai; Hu Hongli; Zhang Yan; Brunzelle Joseph S; Couture Jean Francois; Shilatifard Ali*; Skiniotis Georgios*.Structure and Conformational Dynamics of a COMPASS Histone H3K4 Methyltransferase Complex.Cell, 2018, 174(5): 1117-1126.
16.Koehl Antoine; Hu Hongli*; Maeda Shoji; Zhang Yan; Qu Qianhui; Paggi Joseph M; Latorraca Naomi R; Hilger Daniel; Dawson Roger; Matile Hugues; Schertler Gebhard F X; Granier Sebastien; Weis William I; Dror Ron O; Manglik Aashish*; Skiniotis Georgios*; Kobilka Brian K*.Structure of the mu-opioid receptor-G(i) protein complex.Nature, 2018, 558(7711): 547-552.
17.Ehsan Muhammad; Ghani Lubna; Du Yang; Hariharan Parameswaran; Mortensen Jonas S; Ribeiro Orquidea; Hu Hongli; Skiniotis Georgios; Loland Claus J; Guan Lan; Kobilka Brian K; Byrne Bernadette; Chae Pil Seok*.New penta-saccharide-bearing tripod amphiphiles for membrane protein structure studies.Analyst, 2017, 142(20): 3889-3898.
18.Zhang Ya*n; Sun Bingfa; Feng Dan; Hu Hongli; Chu Matthew; Qu Qianhui; Tarrasch Jeffrey T; Li Shane; Kobilka Tong Sun; Kobilka Brian K; Skiniotis Georgios.Cryo-EM structure of the activated GLP-1 receptor in complex with a G protein.Nature, 2017, 546(7657): 248-253.
19. Shoji Maeda, Antoine Koehl, Hugues Matile, Hongli Hu, Daniel Hilger, Gebhard Schertler, Aashish Manglik, Georgios Skiniotis, Roger Dawson, and Brian Kobilka. “Development of an antibody fragment that stabilizes GPCR/G-protein complexes", Nature Communications 9 (1), 3712 (2018)
20.Hongli Hu; Xing Meng.Observation of Network Dynamics of Ryanodine Receptors on Skeletal Muscle Sarcoplasmic Reticulum Membranes.European Journal of Translational Myology, 2016, 26(2).
21.Hu Hongli; Zhang Kaiming; Meng Xing*.Classification using diffraction patterns for single-particle analysis.Ultramicroscopy, 2016, 164: 46-50.
22.Liu, Zhouying; Du, Xiangning; Deng, Jie; Gu, Mingyue; Hu, Hongli; Gui, Miao; Yin, Chang-Cheng; Chang, Zhenzhan*.The interactions between mitochondria and sarcoplasmic reticulum and the proteome characterization of mitochondrion-associated membrane from rabbit skeletal muscle.Proteomics, 2015, 15(15): 2701-2704.
23.Zhang, Kaiming; Ezemaduka, Anastasia N.; Wang, Zhao; Hu, Hongli; Shi, Xiaodong; Liu, Chuang; Lu, Xinping; Fu, Xinmiao; Chang, Zengyi; Yin, Chang-Cheng*.A Novel Mechanism for Small Heat Shock Proteins to Function as Molecular Chaperones.Scientific Reports, 2015, 5: 8811.
24.Hu, Hongli; Wang, Zhao; Wei, Risheng; Fan, Guizhen; Wang, Qiongling; Zhang, Kaiming; Yin, Chang-Cheng*.The molecular architecture of dihydropyrindine receptor/L-type Ca2+ channel complex.Scientific Reports, 2015, 5: 8370.
荣誉奖励:
1、2020中国新锐科技人物突出成就奖。
专访 | 胡红丽教授:以“镜”制“冻”,用冷冻电镜探索微观世界
2019-06-24
胡红丽博士于2019年6月正式加入香港中文大学(深圳)生命与健康科学学院,担任助理教授、博士生导师及科比尔卡创新药物开发研究院首席研究员。本期采访中,胡教授不仅把她的科研经历娓娓道来,也对冷冻电子显微镜技术进行了介绍。
Q1. 您的学习经历是怎样的?
我本科就读于南开大学应用物理系。南开大学数学系比物理系要强一些,但是我不是很喜欢纯理论纯计算的学科,所以高考报名的时候就优先选择了物理系。应用物理学是一个交叉学科的专业,包括生物、医学、光电子信息科技、光学等细分方向。本科前两年学习了物理学的基础课程之后,到三年级细分专业方向时,我选择的就是生物物理学方向,所以我在本科阶段就对生物学有了一定的知识积累。到大四选择保研还是找工作的时候,我当时想继续从事科研。机缘巧合,北医(北京大学医学部)的尹长城教授来南开进行讲座,他当时讲的就是冷冻电子显微镜的技术,我听了之后很震惊,用冷冻电子显微镜技术居然可以直接“看”到需要研究的生物分子,我觉得这个技术如果发展起来会有非常广泛的应用空间,所以当时毕业设计的时候我就选择进到他的组做实习,本科毕业后也选择了他作为我的博士生导师,正式进入了冷冻电镜这一研究领域。可以说,尹老师是我科研上的领路人。本科阶段对物理学,数学和生物学的交叉学科知识的掌握也帮助了我从事冷冻电镜方面的结构生物学研究。
Q2. 您能简单介绍一下冷冻电镜技术的发展吗?
1931年德国科学家Ernst Ruska发明了透射电子显微镜。上世纪中叶,生物学家尝试对生物样品进行固定、包埋、切片及重金属染色等技术应用电子显微镜观察细胞的精细结构,并发现了许多重要的细胞器。但是这样观察到的生物样品是脱水状态的,而且是对附着生物分子的重金属染料成像,无法获得生物大分子的高分辨结构信息。如何用电子显微镜观察含水状态的生物样品成了科学家们试图解决的问题。
上世纪80年代Jacques Dubochet发明了快速冷冻的方法,可以将生物分子在溶液中的状态迅速固定。当降温速度比较快时,水溶液会形成无序的玻璃态冰,而不是我们常见到的晶体状冰,这样生物大分子可以保持其在溶液中的原始结构,而低温环境可以降低电子对生物大分子的辐射损伤,保持了蛋白质的高分辨结构信息。这种用低温制备并观察样品的技术成为现在普遍使用的冷冻电子显微学技术。Jacques Dubochet也因为自己对冷冻电镜技术发展的原创性贡献,与Joachim Frank和Richard Henderson共同分享了2017年诺贝尔化学奖。
在成像方面,透射电子显微镜的成像是电子束与生物大分子相互作用后的二维投影信息。1968年,Aaron Klug发现了中心截面定理,将采集到的生物样品的多个角度的电子显微图像在计算机中重构出它的三维结构。生物分子在溶液中以随机的取向分布在样品中,当我们对生物分子大量成像后,理论上可以采集到他们在空间各个角度的投影信息。用这种方法获得生物分子三维结构相比于晶体学衍射技术来说有很多优点。首先是不需要对生物分子结晶,我们知道晶体学进行结构研究最大的限制是结晶困难,并非所有生物分子都能形成高度有序的晶体。其次是冷冻电子显微镜是在生物分子的原始溶液中直接观察他们的状态,可以最大程度保持生物分子在溶液中的自然状态。Aaron Klug也因此获得了1982年诺贝尔化学奖。
在我2008年开始读博的时候,冷冻电镜并没有广泛的应用,同年Hong Zhou在Nature上发表了3.3埃分辨率的多角体病毒结构,在当时造成比较大的轰动,首次证明了冷冻电子显微镜可以作为重要技术手段解析生物大分子的原子模型。但是受各种技术水平的限制,它只在研究病毒这一类尺寸比较大、对称性比较高的生物结构时才能够达到较高的分辨率,所以当时冷冻电镜算是比较弱势的学科,我们在北大的时候还经常参加“弱势学科讨论会”。
我作为博士研究生的几年是比较幸运的,见证了冷冻电镜领域最重要的几项技术变革。到今天,冷冻电镜已经和X射线、核磁共振并列为结构生物学的三大技术。在硬件方面,厂家一方面在努力提高电子显微镜的机械系统和光学系统的稳定性,尤其是新开发的三级聚光镜的系统保证了电子束的相干性很好。另外一个就是样品台,之前都是人工操作转移和插入样品,后来发展出来用机械手取代人工负责样品转移和把样品放到操作台上,这样的好处就是更稳定,同时所有操作都与外界隔绝也可以避免样品受到冰的污染。在图像采集方面,基于CMOS技术开发的直接电子探测器取代了CCD和胶片,可以对样品进行高速连续采样,大大的提高了探测效率。在电子束照射下冷冻样品会有一个明显的漂移,称为drift,使用直接电子探测器,我们可以记录非常短的时间内拍摄到的信号,然后对每个时间点的照片上样品的移动进行校正。打个比方就是相机的快门速度和感光元件灵敏度都提高很多,每一张照片都可以理解为一个电影,我们已经可以做到对电影的每一帧进行校正。在软件上,英国剑桥大学的Sjors Scheres开发的单颗粒三维重构分析算法并开发为实用的软件RELION,对样品里面不同状态的颗粒的图片进行二维和三维的分类,有效将不同构象状态的结构通过分类区分,这个软件已成为冷冻电镜结构解析的主流软件包。2013年UCSF(加州大学旧金山分校)的David Agard和Yifan Cheng用冷冻电镜技术解析出来的TRPV1近原子分辨率结构,至此,冷冻电镜进入一个全新的时代,众多过去无法解析的低分子量、无对称的重要生物大分子结构被解析出来。
Q3. 您的科研经历是怎样的?
我在博士期间和博士后前两年做的都是钙通道蛋白的结构解析,一个是钙离子的释放通道,一个是电压门控的钙通道。我用这宝贵的几年时间学习了各种电子显微镜相关的理论知识和实践经验,由于经常需要到其他实验室借用电子显微镜,可以说几乎和所有旧式的电子显微镜都有过接触,打下了坚实的基础。
博士毕业后我在纽约州卫生部下属的Wadsworth Center研究所从事冷冻电镜相关的研究,Wadsworth Center曾经是前面提到的与Jacques Dubochet分享2017年诺贝尔化学奖的Joachim Frank教授的实验室所在地,但是我去的时候他已经搬到纽约哥伦比亚大学了。我在Wadsworth的导师是Frank的学生Terry Wagenknecht,不过他当时也已经到了快退休的年龄,我在那里又学习了冷冻电子显微镜的另一项重要技术:电子断层扫描成像。后来实验室的导师退休,我找工作的时候联系了密歇根大学的Georgios Skiniotis教授,面试的时候他比较看重我的科研经历里面丰富的冷冻电镜的经验。我到了实验室之后才知道他和获得2012年诺贝尔化学奖的两位教授——杜克大学的Robert Lefkowitz教授和斯坦福大学的Brian Kobilka教授——都有密切的合作。
刚进实验室时,我就接手了一个GPCR(G蛋白偶联受体)的复合物的课题。我去之前,实验室的两位博后已经做了一些尝试。这一类复合物分子量比较小,当时认为用冷冻电子显微镜解析结构还比较困难,复合物又非常不稳定,我们在制备冷冻电镜样品的时候一直遇到很多困难,我加入之后就和他们在样品筛选和制备方面做了很多实验,终于取得了一些进展。另一方面,当时世界上仅有少数实验室设置了自动数据采集软件来收集数据,我们还是用手动的方式,每张照片都要操控样品杆把样品移到中心,然后进行聚焦和曝光,往往收集一套数据要重复成千上万次,效率也比较低,即便24小时不休息,一天也只能照几百张照片。我就和导师提出要安装一个自动数据采集软件,把人力解放出来,他同意后我就结合之前学过的知识摸索如何安装和设置自动数据采集软件。到2017年初,我刚把密大的一台冷冻电镜的自动收集程序调试运行成功时,Georgios决定把实验室搬到斯坦福,以便和Kobilka实验室的继续合作。当时斯坦福已经安装了四台冷冻电镜,但是软件和调试工作都没开始。Georgios说服我去斯坦福继续和他一起工作,包括让我负责电镜的软件安装和调试工作,希望把我在电镜技术上的积累和创新充分利用起来。
这几年内我和Kobilka教授的合作有很多,其中有两个成果是最有代表性的。第一个课题是关于GPCR和G蛋白复合物的,我们研究的是阿片受体,阿片受体是镇痛剂的靶点,它是通过结合G蛋白向下游传递信号的。Kobilka课题组对阿片受体的工作机制的研究已经开展了很多年,他们用晶体学的方法分别解析了阿片受体蛋白在失活和激活时的构象,了解了受体激活过程的结构变化,但是我们并不清楚不同的激动剂激活方式的区别以及阿片受体是如何选择性结合G蛋白向下传递信息。当时Kobilka教授的一个博士研究生花了几年时间尝试结晶阿片受体与G蛋白的复合物,拿到了不少晶体,但是都没有衍射信号,所以他找我们合作,希望可以用冷冻电镜方法研究它的结构。这个工作是我在密歇根大学的时候开展的,我一边测试样品一边测试自动收集软件。刚开始的时候,在薄冰中找不到蛋白颗粒,只能在厚冰处收集数据,图像衬度比较差,分辨率也很难提高。后来我们改进了制样技术,把复合物冻到很薄的冰层中,图像信噪比有很明显的提高,最后得到3.5埃分辨率的结构,可以非常精确的看到受体和G蛋白的作用位点。这项成果于2018年发表在Nature杂志上。Nature在同一期发表了另外三篇GPCR复合物结构相关的文章,可见这一领域的竞争是非常激烈的,在同期的四篇论文中,我们的分辨率是最高的。
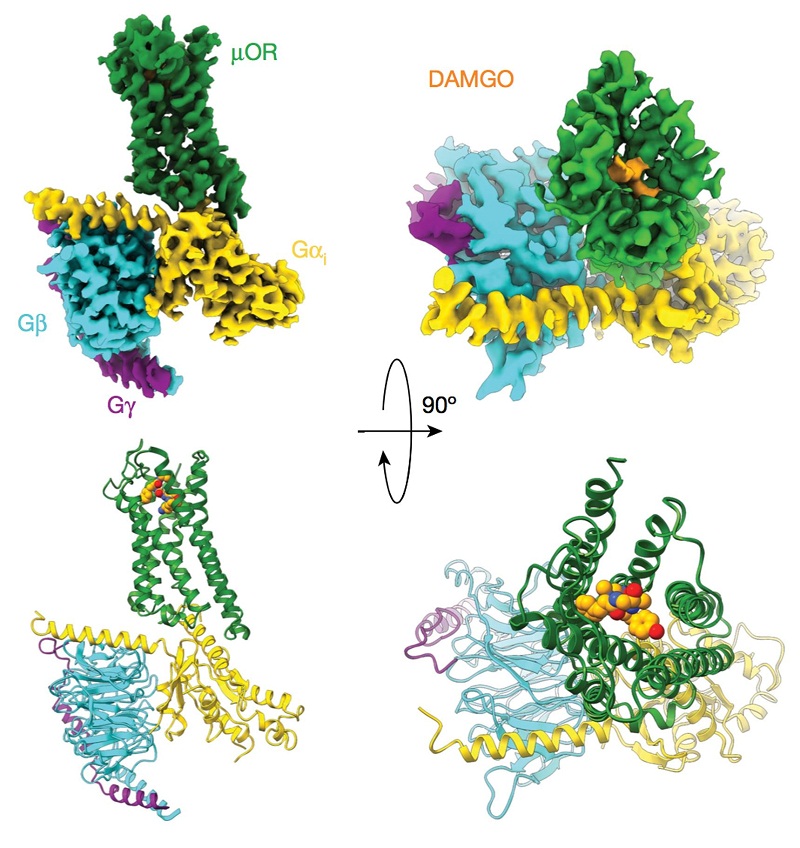
上图为胡教授2018年发表的Nature一作论文插图,原文引用为:Antoine Koehl*, Hongli Hu*, et al, “Structure of the μ Opioid Receptor-Gi Protein Complex”,
Nature, 558(2018):547-552
第二项成果是Class C的GPCR的结构,这项成果也是非常有代表性的,因为C类GPCR和其他类型GPCR差别很大。所有的GPCR都有七次跨膜螺旋,但C类GPCR在胞外有个很大的结构域,而且它的正构位点(orhtosteric site)不在七次跨膜区,而在胞外的结构域上。我们研究的代谢型谷氨酸受体5(简称mGlu5)是参与调节中枢神经系统的突触传递和神经元兴奋性受体,一般是以二聚体的方式行使功能。Kobilka课题组曾经用晶体学的方法做了很多尝试,只能得到胞外区的结构,使用全长的分子进行结晶得到衍射也无法算出跨膜区的结构,也就无法得知配体结合到正构位点之后是如何传递到跨膜区的。我们用冷冻电镜方法分别成功解析了mGlu5在失活和激活状态的两个结构,失活状态胞外VFT(捕蝇器)区域是张开的,二聚体也是几乎平行的。配体结合之后被VFT捕捉,然后VFT关闭,带动二聚体产生一个比较大的位移和旋转,跨膜区也随之距离变短,7次跨膜的构象也发生变化。我们用突变的方法验证了二聚体激活后的结合位点与mGlu5行使功能相关。这是首次观察到完整结构的C类GPCR的构象变化,这项成果在Nature杂志发表后又专门有一篇短讯刊登在同一期杂志News & Reviews,在Twitter上也引起众多转载。
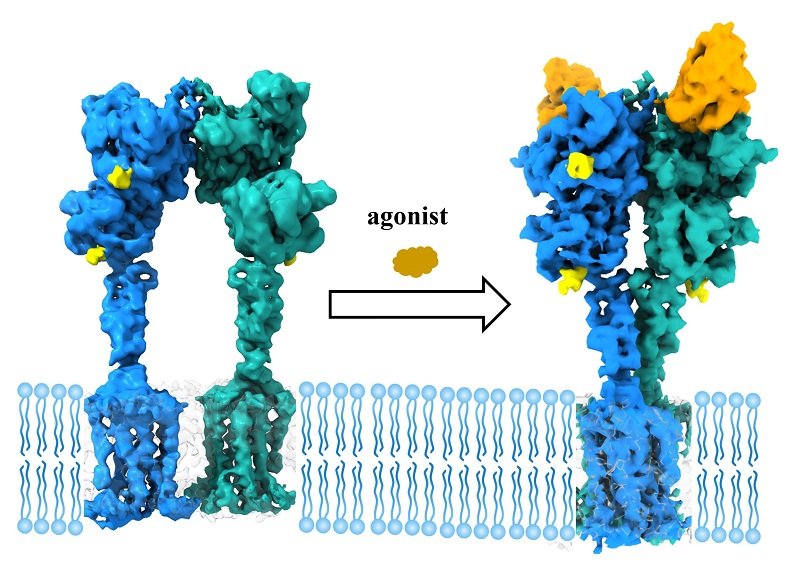
上图为胡教授2019年发表的Nature一作论文插图,原文引用为:Antoine Koehl*, Hongli Hu*, et al "Structural Insights into Metabotropic Glutamate Receptor Activation". Nature, 566(2019):79–84
Q4. 您为什么选择加入港中大(深圳)?
香港中文大学(深圳)的科比尔卡创新药物开发研究院2017年成立的时候,我在美国就关注过,当时我的课题有了一些阶段性的成果,我还憧憬了一下将来可以来到这里工作。去年下半年,我的两个重要成果都发表之后,我开始认真考虑回国发展,第一个就想到了这里,拿到文章接受信之后我就开始准备面试。Kobilka教授和他的夫人Tong Sun都非常支持我,去年11月的时候我就得到了面试的机会。面试过程中和叶院长、朱宝亭教授等学校和学院领导进行了深入的交谈,我也更加深入地了解香港中文大学(深圳),感受到港中大(深圳)欣欣向荣的发展面貌。香港中文大学(深圳)拥有良好的科研氛围和高水平的科研平台,Kobilka研究院购买了两台最高端型号的冷冻电子显微镜Titan Krios,并且配备了强大的计算机集群。学校和学院领导都很重视Kobilka研究院的建设和发展,研究院的几位PI在科研背景上有很好的互补性,可以快速推动科研工作的进展,相信在叶院长和Kobilka教授带领下,我们很快会在药物开发和技术应用方面做出成果。
Q5. 能介绍一下您未来的研究方向吗?
我在GPCR方向的课题有了不错的积累,想继续进行更加深入的研究。GPCR家族有800多个成员,对人体生理代谢的几乎所有方面都有重要影响,现今市场上超过30%的药物都是以这一类蛋白为靶点的,然而到目前为止仅有不到70个GPCR的结构被解析,还有许多具有重要生理学功能的GPCR有待我们去研究。冷冻电镜目前已经是结构生物学研究的主流方式,为我们了解GPCR的结构和功能提供重要支持。一方面,我们要研究GPCR与不同类型配体相互作用的复合物的结构,解析受体在不同功能状态下的构象变化。另一方面,GPCR的下游蛋白并不只有G蛋白,还有β-arrestin和GRK等,GPCR可以通过结合不同的配体激发的构象变化,与不同的下游信号分子相互作用继而引发复杂的信号转导过程,然而特定类型的GPCR如何与激发不同的信号传导还需要更多结构生物学信息提供支持。Kobilka研究院主要从事创新药物开发,冷冻电子显微镜给药物开发提供了非常好的手段,通过冷冻电镜研究蛋白的药物靶点的结构,为药物开发和优化提供更多的依据。
此外,在冷冻电镜技术方面也可以继续进行研究。冷冻电子显微镜的应用越来越广泛,但是它还是有一定的技术门槛的,在样品制备、数据采集和数据处理环节中,操作人员都需要受过一定时间的专业培训,结构解析过程也需要科研人员在三维重构、编程和生物学方向的专业知识。将来我们可以在方法学上进行研究,优化现有的操作流程和方法,使得这项技术发展的更加成熟,降低技术门槛,让越来越多的科研人员能够娴熟地应用冷冻电镜技术。
Q6. 您对我们的学生有哪些建议?
在对待科研的态度方面,我自己的感觉就是,兴趣最重要。科研在外人看来还是比较“高大上”的,做科研每天接触到的都是最前沿的知识,每当有新的成果做出来或者发现新的现象,激动的心情都是无法用言语表达的。然而在实际工作中,大部分时间要应对许多枯燥、辛苦的工作,经常需要阅读大量的文献才能找到某个问题的答案,没有兴趣很难坚持下来。另外,科研工作的研究结果往往是未知的,并非所有结果都能达到预期,或者在实验过程中经常遇到瓶颈长期没有进展,需要有很大的毅力才能坚持下来。
至于同学们专业的选择,除了兴趣以外,也要充分了解专业的发展趋势,要看学科和专业的未来会有怎样的发展。多和业内的人士沟通,来判断是否真的是自己想要做的方向。在现在这个信息化的社会,许多工作都需要各个行业的人才紧密合作才能完成,无论选择什么方向都要重视和加强人际间合作交流的能力。
人物简介

胡红丽博士于2008年获得南开大学应用物理学专业学士学位,2013年获得北京大学医学部生物物理学专业博士学位,随后赴美国开展博士后的研究工作。分别在美国纽约州卫生部Wadsworth Center、密歇根大学和斯坦福大学从事基于冷冻电子显微镜的结构生物学的研究,其中在密歇根大学和斯坦福大学期间与2012 年诺贝尔化学奖得主 Brian K. Kobilka 教授和Robert J. Lefkowitz教授展开广泛的合作研究,参与了许多重要项目,在解析G 蛋白偶联受体(简称 GPCR)高分辨的复合物结构方面做出了突出贡献。胡红丽博士自2019年6月起入职香港中文大学(深圳)生命与健康科学学院及科比尔卡创新药物开发研究院担任助理教授/独立PI、博士生导师。
胡红丽博士主要从事 GPCR的复合物研究,运用冷冻电镜技术研究得到 GPCR 的高分辨结构,并研究膜蛋白与其调控蛋白之间相互作用,为设计以膜蛋白为靶标的药物提供重要的理论依据。自2017年起在 Nature、Cell 等顶级杂志上发表文章五篇,其中以共同第一作者身份在Nature杂志发表文章两篇。主要成果包括:阿片受体与抑制型G蛋白Gi 的3.5埃高分辨电镜结构,以及首个C类GPCR代谢型谷氨酸受体(mGlu5)全长分子不同状态的电镜结构,两个结构均发表在Nature杂志上,两篇文章均被Nature News & Review着重报道。
中国科技创新人物云平台暨“互联网+”科技创新人物开放共享平台(简称:中国科技创新人物云平台)免责声明:
1、中国科技创新人物云平台是:“互联网+科技创新人物”的大型云平台,平台主要发挥互联网在生产要素配置中的优化和集成作用,将互联网与科技创新人物的创新成果深度融合于经济社会各领域之中,提升实体经济的创新力和生产力,形成更广泛的以互联网为基础设施和实现工具的经济发展新形态,实现融合创新,为大众创业,万众创新提供智力支持,为产业智能化提供支撑,加快形成经济发展新动能,促进国民经济提质增效升级。
2、中国科技创新人物云平台暨“互联网+”科技创新人物开放共享平台内容来源于互联网,信息都是采用计算机手段与相关数据库信息自动匹配提取数据生成,并不意味着赞同其观点或证实其内容的真实性,如果发现信息存在错误或者偏差,欢迎随时与我们联系,以便进行更新完善。
3、如果您认为本词条还有待完善,请编辑词条。
4、如果发现中国科技创新人物云平台提供的内容有误或转载稿涉及版权等问题,请及时向本站反馈,网站编辑部邮箱:kjcxac@126.com。
5、中国科技创新人物云平台建设中尽最大努力保证数据的真实可靠,但由于一些信息难于确认不可避免产生错误。因此,平台信息仅供参考,对于使用平台信息而引起的任何争议,平台概不承担任何责任。

















